Real-time PCR- ilościowa reakcja łańcuchowa polimerazy
Real-time PCR to inaczej PCR w czasie rzeczywistym, bądź, jak nazwa wskazuje, ilościowy PCR (ang. quantitative PCR, qPCR), który pozwala na określenie ilości danej sekwencji w próbie za pomocą pomiarów fluorescencji.
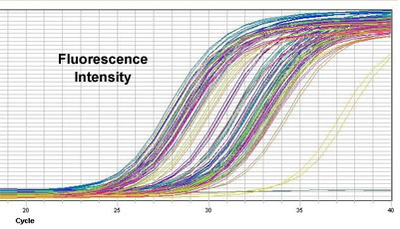
Metoda ta umożliwia nam wgląd w przebieg całej reakcji PCR poczynając od fazy wstępnej, przez fazę wzrostu logarytmicznego aż do fazy plateau (Rys. 1). W fazie wzrostu logarytmicznego fluorescencja próbki przekracza fluorescencję tła (w tzw. cyklu progowym) co pozwala na wiarygodne monitorowanie fluorescencji. Próbka tym szybciej osiągnie wartość progową fluorescencji (ang. fluorescence treshold) im większe było stężenie wyjściowe powielanej sekwencji. Aby określić stężenie wyjściowe produktu należy wyznaczyć punkt przecięcia się linii progowej a także wyznaczyć krzywą standardową punktów przecięcia dla szeregu stężeń standardów.
Dzięki zastosowaniu fluorescencji, Real-Time PCR charakteryzuje się znacznie lepszą czułością i precyzją w porównaniu do standardowego PCR. Umożliwia to oznaczanie nawet przy niewielkim stężeniu produktu. Możliwa jest zarówno analiza jakościowa oraz ilościowa. Dodatkową zaletą tej metody jest zmniejszenie ilości czasu potrzebnego do jej wykonania. Rekcja zachodzi w jednej probówce, w której wykonywane są pomiary fluorescencji w czasie rzeczywistym pozwalające na określenie ilości produktu. Metoda ta charakteryzuje się również wysoką powtarzalnością. Wizualizację fluorescencyjną przyrastającego produktu można wykonać stosując barwniki interkalujące DNA. Ogranicza to koszty, jednak jest niespecyficzne, gdyż barwniki te wiążą się do każdej cząsteczki DNA. Barwniki najczęściej stosowane to: SYBR Green.
Drugą metodą oznaczania jest zastosowanie sond fluorescencyjnych czyli znakowanych fluorescencyjnie nukleotydów. Metoda ta jest znacznie droższa od poprzedniej gdyż wymaga zaprojektowania odpowiednich sond, jednak jej zaletą jest wysoka specyficzność, gdyż sondy te są komplementarne do powielanej sekwencji. Przy projektowaniu sond należy jednak pamiętać o tym, że ich temperatura topnienia musi być przynajmniej o 10°C wyższa od temperatury topnienia starterów. Jeśli tego się nie uwzględni starter może zablokować miejsce łączenia się sondy. Najczęściej stosowanymi sondami są:
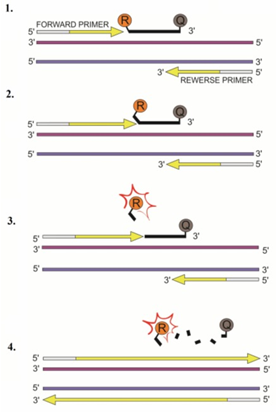
-TaqMan - które posiadają na końcu 5’ barwnik reporterowi a na końcu 3’ barwnik wygaszający. Dzięki temu sondy, które się nie przyłączyły do cząsteczki DNA nie będą dawać fałszywego sygnału. Sygnał fluorescencyjny uzyskiwany jest dzięki aktywności 5’-3’ egzonukleazowej polimerazy, która hydrolizuje sondę.
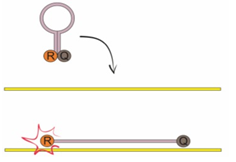
-Molecular Beacon - sondy te tworzą strukturę spinki do włosów dzięki temu, że posiadają specjalne komplementarne do siebie sekwencje flankujące. Tak jak w przypadku TaqMan, obecny jest barwnik reporterowy oraz barwnik wygaszający, każdy na jednym z fragmentów flankujących. Sonda przyłącza się i jednocześnie rozplata, tylko w przypadku napotkania idealnie komplementarnej sekwencji. Takie rozwiązanie powoduje, że sondy te są niezwykle czułe.
-Sondy Skorpion –sondy te są modyfikacją Molecular Beacon. Do struktury spinki dołączony jest starter. W swojej budowie jest dość skomplikowana gdyż dodatkowo posiada element blokujący polimerazę znajdujący się między końcem 5’ startera i końcem 3’ sondy, gdzie zlokalizowany jest również wygaszacz. Barwnik fluorescencyjny znajduje się na końcu 3’ sondy. Wolny pozostaje koniec 3’ startera, który pozwala na przyłączenie się polimerazy.
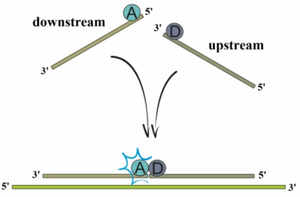
-Liniowe sondy oligonukleotydowe –w tej metodzie wykorzystywane są dwie sondy, downstream i upstream, z barwnikami fluorescencyjnymi, z których jeden jest donorem a drugi akceptorem fluorescencji. Powoduje to, że obserwuje się emisję akceptora jedynie gdy sondy te zbliżą się do siebie na matrycy DNA. Sonda downstream posiada barwnik na końcu 5’ natomiast upstream na końcu 3’.
Literatura
Turner P., McLennan A., Bates A., White M. Molecular Biology. Instant Notes. Third Edition (2005), Taylor & Francis Group. ISBN 0-415-35167-7
Schleif R. Genetics and Molecular Biology. Second Edition (1993). The John Hopkins University Press. ISBN 0-8018-4673-0
Khot P.D., Fredrick D.N. PCR-based diagnosis of human fungal infecions. Expert Review of Anti-infective Therapy 2009: 7(10): 1201-1221
McPherson M., Moller S. PCR. The BASICS. Second Edition (2006), Taylor & Francis Group. ISBN 0-4153-5547-8
Ilustracje:
http://www.assay-protocol.com/molecular-biology/real-time-pcr