(→POLIMERAZA) |
(→PRIMERY (STARTERY)) |
||
| Linia 31: | Linia 31: | ||
Enzymy mogą się różnić posiadaniem właściwości endonukleazy 3’->5’, czyli również dokładnością czy termostabilością. Ważnym jest aby poprawnie dobrać polimerazę do planowanego eksperymentu. | Enzymy mogą się różnić posiadaniem właściwości endonukleazy 3’->5’, czyli również dokładnością czy termostabilością. Ważnym jest aby poprawnie dobrać polimerazę do planowanego eksperymentu. | ||
| − | == | + | ==Primery (Startery)== |
Startery są bardzo ważnym elementem PCR, należy je odpowiednio zaprojektować aby uzyskać pożądany efekt, czyli kopie danej sekwencji. Dodatkowo, ze względu na to, że próbka DNA zawiera cząsteczki dłuższe niż interesująca nas sekwencja, startery zapewniają syntezę jedynie fragmentu, który ma być powielony. Można powiedzieć, że polimeraza dzięki nim „wie” co ma być zsyntetyzowane. | Startery są bardzo ważnym elementem PCR, należy je odpowiednio zaprojektować aby uzyskać pożądany efekt, czyli kopie danej sekwencji. Dodatkowo, ze względu na to, że próbka DNA zawiera cząsteczki dłuższe niż interesująca nas sekwencja, startery zapewniają syntezę jedynie fragmentu, który ma być powielony. Można powiedzieć, że polimeraza dzięki nim „wie” co ma być zsyntetyzowane. | ||
| Linia 37: | Linia 37: | ||
Przed przystąpieniem do projektowania starterów należy znaleźć taką sekwencję 18-30 nt, która będzie zawierać odpowiednią ilość zasad G, C oraz będzie w miarę możliwości mało powtarzalna, tak by startery nie przyłączyły się niespecyficznie. Istotne również aby oba startery przyłączały się w tej samej temperaturze, co zapewnia jednoczesną syntezę obu nici DNA. | Przed przystąpieniem do projektowania starterów należy znaleźć taką sekwencję 18-30 nt, która będzie zawierać odpowiednią ilość zasad G, C oraz będzie w miarę możliwości mało powtarzalna, tak by startery nie przyłączyły się niespecyficznie. Istotne również aby oba startery przyłączały się w tej samej temperaturze, co zapewnia jednoczesną syntezę obu nici DNA. | ||
| − | Często w reakcji PCR używa się również primerów zdegenerowanych. Tworzy się je gdy sekwencja danego regionu DNA nie jest znana, lub znana jest tylko część. Projektuje się wtedy kilka rodzajów primerów, gdzie każdy z nich zawiera inny kod danego aminokwasu. | + | Często w reakcji PCR używa się również primerów zdegenerowanych. Tworzy się je gdy sekwencja danego regionu DNA nie jest znana, lub znana jest tylko część. Projektuje się wtedy kilka rodzajów primerów, gdzie każdy z nich zawiera inny kod danego aminokwasu. |
==CYKLE== | ==CYKLE== | ||
Wersja z 20:34, 11 lis 2014
Spis treści
PCR - Łańcuchowa Reakcja Polimerazy
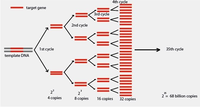
Łańcuchowa Reakcja Polimerazy (ang. polymerase chain reaction) to metoda in vitro powszechnie stosowana w biologii i medycynie molekularnej. Podstawową zasadą tej metody jest powielanie określonej sekwencji DNA (Rys. 1) w niewielkiej probówce. Opisana i zastosowana po raz pierwszy w 1985 roku przez Saiki et. al. (1985).
Technika ta pozwala na powielenie specyficznej sekwencji, która znajduje się w centrum zainteresowania. W tym celu tworzone są tak zwane primery (startery). Są to jednoniciowe sekwencje DNA, składające się z ok 18-30 nukleotydów. Każda z nich jest komplementarna do jednego z końców wspomnianej sekwencji DNA. Po połączeniu się primerów z sekwencją DNA polimeraza DNA wydłuża każdy ze starterów dobudowując drugą nić DNA na nici matrycowej, do której dołączył się starter.
Łańcuchowa reakcja polimerazy przeprowadzana jest w specjalnych, niewielkich probówkach (o pojemności 200µl), które układane są w bloku grzejnym - termocyklerze (Rys.2), zaprogramowanym na wykonanie odpowiedniej ilości cykli. W mieszaninie reakcyjnej powinny się znaleźć nukleotydy i startery, które powinny być dodane w nadmiarze, próbka DNA, polimeraza, oraz bufor reakcyjny, zawierający jony magnezu, które potrzebne są do prawidłowej pracy polimerazy.
Metoda PCR jest niezwykle czułą techniką, która pozwala na wykrycie zaledwie jednej cząsteczki, także przy obecności nawet 106 innych. Stosowana jest między innymi w sekwencjonowaniu, pozwala na identyfikację mutacji bądź defektów genetycznych. Ponadto stosowana jest w określaniu obecności markerów nowotworowych, obecności wirusa HIV czy infekcji grzybiczych.
Ówcześnie w laboratorium stosuje się różne odmiany reakcji PCR, niektóre z nich to:
-RT-PCR (reverse transcription PCR)
-Real-time-PCE lub qPCR (quantitative PCR)
-qRT-PCR
-nested PCR
-multiplex PCR i inne
Polimeraza
Polimeraza jest enzymem, który powiela nici DNA. W reakcji PCR, ze względu na wysokie temperatury jakie są wykorzystywane podczas reakcji używa się termostabilnych polimeraz wyizolowanych z bakterii lub Archeae żyjących w wysokich temperaturach. Najpopularniejszą jest polimeraza Taq pochodzącą z Termusaquaticus. Wadą jest jednak brak właściwości naprawczych co powoduje tworzenie błędów przez ten enzym raz na około 250 nukleotydów. Oczywiście dostępne są różne ulepszone wersje tego enzymu tworzone przez firmy biotechnologiczne lub inne polimerazy, które są bardziej dokładne np. polimeraza Pfu z Pyrococcusfuriosus, która posiada właściwości endonukleazy 3’->5’. Kolejnymi polimerazami używanymi w reakcji PCR to np. Pab-polB z Pyrococcusabyssi lub Tma-polA z Thermotogamaritima.
Enzymy mogą się różnić posiadaniem właściwości endonukleazy 3’->5’, czyli również dokładnością czy termostabilością. Ważnym jest aby poprawnie dobrać polimerazę do planowanego eksperymentu.
Primery (Startery)
Startery są bardzo ważnym elementem PCR, należy je odpowiednio zaprojektować aby uzyskać pożądany efekt, czyli kopie danej sekwencji. Dodatkowo, ze względu na to, że próbka DNA zawiera cząsteczki dłuższe niż interesująca nas sekwencja, startery zapewniają syntezę jedynie fragmentu, który ma być powielony. Można powiedzieć, że polimeraza dzięki nim „wie” co ma być zsyntetyzowane.
Przed przystąpieniem do projektowania starterów należy znaleźć taką sekwencję 18-30 nt, która będzie zawierać odpowiednią ilość zasad G, C oraz będzie w miarę możliwości mało powtarzalna, tak by startery nie przyłączyły się niespecyficznie. Istotne również aby oba startery przyłączały się w tej samej temperaturze, co zapewnia jednoczesną syntezę obu nici DNA.
Często w reakcji PCR używa się również primerów zdegenerowanych. Tworzy się je gdy sekwencja danego regionu DNA nie jest znana, lub znana jest tylko część. Projektuje się wtedy kilka rodzajów primerów, gdzie każdy z nich zawiera inny kod danego aminokwasu.
CYKLE
W rekacji PCR powtarza się po kilkadziesiąt razy cykle, które złożone są z kilku etapów, w których znacznie zmieniana jest temperatura. Pierwszym z nich jest denaturacja, która pozwala na rozdział podwójnej nici DNA. To w następnym etapie- etapie przyłączania- pozwala na połączenie się starterów z matrycą. Następnym krokiem jest wydłużanie, czyli replikacja DNA. W tym etapie bierze udział polimeraza. Następnie cały cykl, od etapu denaturacji jest powtarzany.
Każdy krok PCR różni się temperaturą w jakiej się odbywa. Denaturacja zachodzi w 95°C, przyłączanie w ok. 55°C, choć ta temperatura może się różnić w zależności od zawartości zasad C i G. Dla etapu wydłużania jest to ok. 72°C, choć często trzeba temperaturę dostosować do warunków optymalnych dla danej polimerazy.
W każdym cyklu z pojedynczej nici DNA powstaje kolejna, dwuniciowa cząsteczka DNA, co oznacza, że przyrost kopii genu jest wykładniczy (Rys.1).
KINTETYKA REAKCJ
Reakcja PCR posiada trzy charakterystyczne fazy cyklu.
- Faza początkowa - gdzie kopie sekwencji wolno narastają, ze względu na to, że jest ich mało, a startery „szukają” odpowiedniego miejsca na przyłączenie się.
- Faza szybkiego wzrostu – jest to faza, w której ilość kopii danej sekwencji narasta w sposób wykładniczy.
- Faza plateau- faza, w której wzrost jest już niewielki, głównie ze względu na czynniki limitujące, takie jak stabilność polimerazy.
ANALIZA PRODUKTÓW PCR
Obecność kopi genu możne być zdeterminowana na różne sposoby. Zwykle stosuje się:
-Elektroforezę w żelu agarozowym,
-Southern blot,
-Dotblot,
-Nested PCR,
-Analizę restrykcyjną,
-Sekwencjonowanie.
Literatura
Turner P., McLennan A., Bates A., White M. Molecular Biology. Instant Notes. Third Edition (2005), Taylor & Francis Group. ISBN 0-415-35167-7.
Schleif R. Genetics and Molecular Biology. Second Edition (1993). The John Hopkins University Press. ISBN 0-8018-4673-0.
Khot P.D., Fredrick D.N. PCR-based diagnosis of human fungal infecions. Expert Review of Anti-infective Therapy 2009: 7(10): 1201-1221.
McPherson M., Moller S. PCR. The BASICS. Second Edition (2006), Taylor & Francis Group. ISBN 0-4153-5547-8.
Ilustracje:
Rysunek 1. : http://www.paulvanouse.com/dwpcr.htmlpcr
Rysunek 2. : https://www.bioke.com/Products/New%20products/suppliers/SensoQuesttermocycler