(→Dokowanie liganda z wykorzystaniem procedury AutoDock.) |
(→Dokowanie liganda z wykorzystaniem procedury AutoDock.) |
||
| Linia 66: | Linia 66: | ||
# Zapisz plik DPF.dpf: | # Zapisz plik DPF.dpf: | ||
#*Docking > Output > Lamarckian GA > DPF.dpf <br><pre style="display;">xsxd dfsdf</pre><br> | #*Docking > Output > Lamarckian GA > DPF.dpf <br><pre style="display;">xsxd dfsdf</pre><br> | ||
| − | |||
# Zadokuj ligand korzystając z procedury AutoDock: | # Zadokuj ligand korzystając z procedury AutoDock: | ||
#*Run > Run AutoDock > (określ katalog w którym znajdują się wszystkie pliki oraz katalog w którym został zainstalowany program AutoDock4.exe) | #*Run > Run AutoDock > (określ katalog w którym znajdują się wszystkie pliki oraz katalog w którym został zainstalowany program AutoDock4.exe) | ||
==Analiza otrzymanych wyników== | ==Analiza otrzymanych wyników== | ||
Wersja z 19:41, 1 cze 2012
Spis treści
- 1 Ćwiczenie: Dokowanie Ligandów
- 2 Przygotowanie receptora oraz liganda.
- 3 Instalacja programu AutoDock oraz ADT (AutoDockTools).
- 4 Wczytanie struktury receptora i liganda.
- 5 Obliczenie siatki potencjałów z zastosowaniem procedury AutoGrid.
- 6 Dokowanie liganda z wykorzystaniem procedury AutoDock.
- 7 Analiza otrzymanych wyników
Ćwiczenie: Dokowanie Ligandów
Celem ćwiczenia jest zbadanie sposobów wiązania się carazololu (typowego antagonisty receptorów adrenergicznych) do receptora β2-adrenergiczneg z zastosowaniem programu AutoDock. Receptory adrenergiczne, podobnie jak rodopsyna, należą do rodziny A receptorów GPCR. Receptory te zlokalizowane są w dużej liczbie tkanek i narządów człowieka, między innymi w centralnym układzie nerwowym, wątrobie, trzustce, nerkach, płytkach krwi i w oku. Typowymi endogennymi ligandami tych białek są hormony, takie jak adrenalina (epinefryna) oraz noradrenalina (norepinefryna). Receptory adrenergiczne biorą udział w kaskadach sygnalizacyjnych: regulujących poziom ciśnienia tętniczego, odpowiadających za częstość skurczów serca, wpływających na funkcje oddechowe oraz mobilizację organizmu do obrony. Receptory adrenergiczne dzielimy na dwa typy α i β. Ponadto w każdej z rodzin, występuje kilka podtypów danego receptora, które różnią się między sobą lokalizacją w organizmie, selektywnością w wiązaniu ligandów, a także sposobem przekazywania sygnału przez białka G.
Poniższe ćwiczenie pokazuje od strony praktycznej jak do miejsca wiążącego białka można zadokować ligand korzystając z progarmu AutoDock oraz pakietu ADT (AutoDockTools):
Przygotowanie receptora oraz liganda.
Strukturę receptora pobrać można z bazy PDB (ang. Protein Data Bank), PDB ID: 2RH1. W pobranym pliku 2RH1.pdb oprócz innych cząsteczek zapisana jest struktura przestrzenna receptora β2-adrenergicznego.
- Strukturę samego receptora należy zachować w osobnym pliku receptor.pdb (reszty od ASP29 do LEU342)
- Współrzedne liganda do pobrania: receptor
Instalacja programu AutoDock oraz ADT (AutoDockTools).
- Program AutoDock należy pobrać ze strony: http://autodock.scripps.edu/downloads a następnie zainstalować go zgodnie z instrukcją
- Program ADT należy pobrać ze strony: http://autodock.scripps.edu/downloads/resources/adt/index_html a następnie zainstalować go zgodnie z instrukcją.
Wczytanie struktury receptora i liganda.
- Należy stworzyć folder (np.: .\dokowanie)w którym będziemy przechowywać wszystkie pliki wymagane przez program AutoDock i ADT a następnie przekopiować tam nasze dwa pliki receptor.pdb oraz ligand.pdb:
- Uruchom program ADT umożliwiający przygotowanie wszystkich potrzebnych plików do programów autogrid4.exe oraz autodock4.exe
- W programie ADT określ folder w którym będą przechowywane wszystkie pliki:
- File > Preferences > Modify Defaults (następnie określ lokalizację stworzonego katalogu z plikami np.: C:\dokowanie)
- Wczytaj strukturę liganda:
- Ligand > Input > Open > ligand.pdb
- Dodaj polarne wodory do liganda. W tym celu należy najpierw zmienić typ atomu azotu znajdującego się w sąsiedztwie grupy –OH liganda:
- Edit > Atoms > Edit Type > (tu należy kliknąć na atom azotu i zmienić jego typ z „Npl” na „N3+”)
- Następnie dodaj polarne wodory:
- Edit > Hydrogens > Add > Polar Only
- Zachowaj strukturę liganda z dodanymi wodorami:
- File > Save > Write PDB > (zachowaj jako ligand_h.pdb)
- Skasuj ligand:
- Edit > Delete > Delete All Molecules
- Wczytaj strukturę ligand z dodanymi wodorami:
- Ligand > Input > Open > ligand_h.pdb
- Określ wiązania ruchome liganda:
- Ligand > Torsion Tree > Detect Root
- Zapisz ligand w formacie *.pdbqt:
- Ligand > Output > Save as PDBQT > ligand.pdbqt
- Wczytanie receptora:
- File > Read Macromolecule > receptor.pdb
- Dodaj polarne wodory do receptora:
- Edit > Hydrogens > Add > Polar Only
- Następnie zapisz receptor w formacie PDBQT:
- Flexible Residues > Chose Macromolecule > receptor.pdb
- File > Save > Write PDBQT > receptor.pdbqt
- Edit > Delete > Delete All Molecules
Obliczenie siatki potencjałów z zastosowaniem procedury AutoGrid.
AutoGrid tworzy trójwymiarową siatkę obejmującą obszar miejsca wiążącego białka. Następnie, atom próbkujący umieszczany jest w każdym węźle siatki. Energia oddziaływania danego atomu z białkiem jest zapisywana kolejno na każdym węźle siatki. Procedura powtarzana jest dla każdego z typów atomów obecnych w ligandzie. W ten sposób tworzone są siatki potencjałów dla wszystkich typów atomów liganda, siatka potencjałów elektrostatycznych oraz siatka potencjałów opisująca energię desolwatacji. Tak otrzymane mapy potencjałów są następnie wykorzystywane przez program AutoDock do oszacowania energii wiązania liganda
- Wczytaj strukturę receptora i liganda w formacie PDBQT:
- Grid > Macromolecule > Open > receptor.pdbqt
- Grid > Set Map Types > Open Ligand > ligand.pdbqt
- Określ dla jakich typów atomów będą liczone siatki:
- Grid > Set Map Types > Directly > Accept
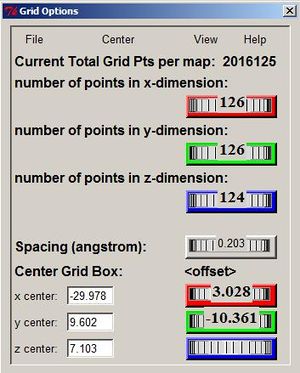
- Zdefiniuj położenie i rozmiar siatki. Miejsce wiązania ligandów w receptorach GPCR znajduje się we wnęce między helisami. Należy tak dobrać parametry siatki aby pokrywała ona całe miejsce wiążące receptora. Nie zapomnij zachować parametrów siatki w oknie Grid Options:
- Grid Box > (przykładowe parametry siatki widoczne na obrazku).
- File > Close saving current
- Zachowaj plik GPF.gpf:
- Grid > Output > Save GPF > GPF.gpf
- Uruchom program AutoGrid:
- Run > Run AutoGrid > (określ katalog w którym znajdują się wszystkie pliki oraz katalog w którym został zainstalowany program AutoGrid4.exe)
Dokowanie liganda z wykorzystaniem procedury AutoDock.
Aby wykonać dokowanie z zastosowaniem procedury AutoDock należy przygotować plik DPF.dpf z parametrami określającymi procedurę dokowania.
- Określ strukture receptora:
- Docking > Macromolecule > Set Rigid Filename > receptor.pdbqt
- Określ strukturę liganda:
- Docking > Choose > ligand > Accept
- Zapisz plik DPF.dpf:
- Docking > Output > Lamarckian GA > DPF.dpf
xsxd dfsdf
- Docking > Output > Lamarckian GA > DPF.dpf
- Zadokuj ligand korzystając z procedury AutoDock:
- Run > Run AutoDock > (określ katalog w którym znajdują się wszystkie pliki oraz katalog w którym został zainstalowany program AutoDock4.exe)