Sekwencjonowanie
Sekwencjonowanie - technika biologii molekularnej polegająca na odczytywaniu kolejności nukleotydów w cząsteczce DNA.
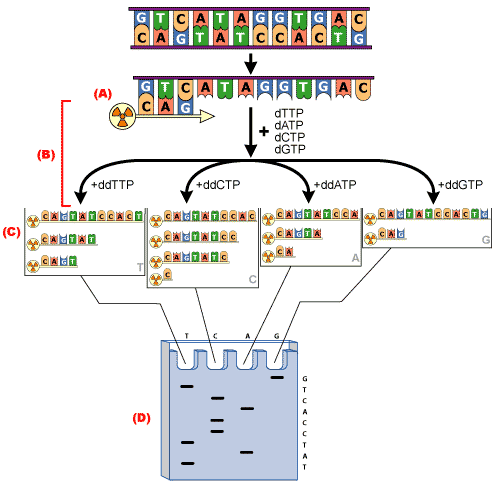
Historycznie, metoda sekwencjonowania, oparta na syntezie DNA z udziałem polimerazy DNA, została opracowana przez Sanger’a w 1977 roku. W metodzie Sanger’a mieszaninę reakcyjną dzieli się na 4 odrębne probówki a do każdej dodaje inny dNTP – dATP, dTTP, DTP, DTP. Poza dNTP do reakcji dodaje się niewielką ilość dideoksynukleotydów ddNTP –odpowiednio ddATP, ddTTP, ddCTP, ddGTP, które są wbudowywane w syntetyzowaną nić DNA. Dołączenie ddNTP uniemożliwia jednak dalsze wydłużanie nici. W ten sposób w powstaje mieszanina fragmentów DNA o różnej długości, zakończonych specyficznym nukleotydem. Mieszaninę produktów rozdziela się następnie elektroforetycznie na żelupoliakrylamidowym i odczytując od dołu rozdziału, ustala sekwencję powstałej nici DNA. Schematyczna ilustracja sekwencjonowania metodą Sanger’a jest przedstawiona na rysunku 1.
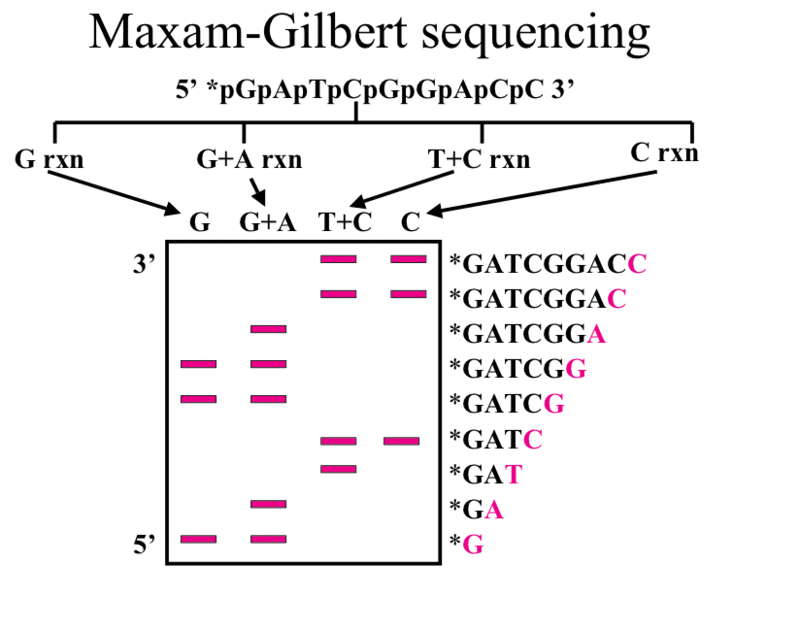
Drugą, alternatywną metodą sekwencjonowania DNA jest metoda chemicznej degradacji Maxama-Gilberta. Metoda ta opiera się na wyznakowaniu promieniotwórczym izotopem 5’ końca DNA a następnie degradacji cząsteczki w 4 odrębnych reakcjach z wykorzystaniem 4 mieszanin odczynników. W pierwszym etapie reakcji odbywa się chemiczna modyfikacja zasad azotowych danego nukleotydu. W miejscu pozbawionym zasad nić ulega pęknięciu za zmodyfikowanym nukleotydem. Do modyfikacji G (guanina) stosuje się mieszaninę piperydyny i siarczanu dimetylu, dla par A+G (adenina i guanina) – siarczan dimetylu, piperydynę oraz kwas mrówkowy, dla C (cytozyna) – hydrazynę/chlorek sodu i piperydynę oraz dla C+T (cytozyna i tymina) – hydrazynę i piperydynę. W wyniku reakcji chemicznej powstają fragmenty DNA wyznakowane na 5’ końcu. Ponieważ dana zasada występuje w łańcuchu wiele razy, w probówce otrzymujemy mieszaninę wielu fragmentów o różnej długości. Podobnie jak w metodzie Sanger’a, fragmenty z czterech oddzielnych reakcji rozdziela się elektroforetycznie na żelu i przy pomocy autoradiografii uwidacznia łańcuchy DNA. Uwidocznione prążki odpowiadają fragmentom DNA posiadającym na 3’ końcu określoną zasadę. Schematyczna ilustracja metody jest przedstawiona na rysunku 2.
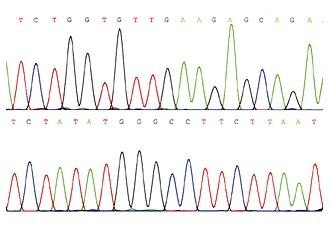
Najpowszechniej obecnie wykorzystywane sekwencjonowanie automatyczne, opiera się na znakowaniu fluorescencyjnym. Każdy z dNTP jest znakowany innym fluorochromem, który ma odmienną falę wzbudzenia. Dzięki temu, iż detektor posiada zdolność rozróżnienia poszczególnych znaczników fluorescencyjnych można przeprowadzić elektroforezę na jednej ścieżce żelu poliakrylamidowego a sekwencja może być odczytywana bezpośrednio w trakcie trwania rozdziału. Wynik jest przedstawiany przez program w postaci 4-kolorowego chromatogramu DNA, gdzie każdy pik reprezentuje zasadę DNA. Przykład wyniku sekwencjonowania ilustruje rysunek 3.