(Utworzył nową stronę „==Proteasom== Proteasom jest złożonym kompleksem białkowym, z czego różne jego elementy mogą wykazywać odmienne właściwości enzymatyczne. Początkowo okre...”) |
|||
| Linia 4: | Linia 4: | ||
Proteasom 26S (o masie 1500-2000 kDa) jest złożony z rdzenia katalitycznego 20S (700 kDa, core particie – CP) i kompleksu regulatorowego 19S (regulatory particie –RP, activator PA700). Proteasom 20S jest zbudowany z 4 pierścieni, które tworzą strukturę cylindryczną. Każdy pierścień z kolei składa się z 7 różnych podjednostek, których masa cząsteczkowa waha się pomiędzy 20 i 35 kDa. Zewnętrzne pierścienie są zbudowane z 7 podjednostek alfa (alfa 1- 7), a dwa pierścienie wewnętrzne z 7 różnych podjednostek beta (beta 1-7). Połączenie pomiędzy podjednostkami beta utrzymuje beta7. NH2-końcowe fragmenty łańcuchów podjednostek alfa zamykają kanał wiodący do wnętrza proteasomu (tzw. alfa-bramka). Otwarcie kanału i aktywacji proteasomu dochodzi na skutek połaczenia z aktywatorem, lub też pod wpływem związków chemicznych. Schematyczna ilustracja budowy proteasomu 26S jest przedstawiona na rysunku 1. | Proteasom 26S (o masie 1500-2000 kDa) jest złożony z rdzenia katalitycznego 20S (700 kDa, core particie – CP) i kompleksu regulatorowego 19S (regulatory particie –RP, activator PA700). Proteasom 20S jest zbudowany z 4 pierścieni, które tworzą strukturę cylindryczną. Każdy pierścień z kolei składa się z 7 różnych podjednostek, których masa cząsteczkowa waha się pomiędzy 20 i 35 kDa. Zewnętrzne pierścienie są zbudowane z 7 podjednostek alfa (alfa 1- 7), a dwa pierścienie wewnętrzne z 7 różnych podjednostek beta (beta 1-7). Połączenie pomiędzy podjednostkami beta utrzymuje beta7. NH2-końcowe fragmenty łańcuchów podjednostek alfa zamykają kanał wiodący do wnętrza proteasomu (tzw. alfa-bramka). Otwarcie kanału i aktywacji proteasomu dochodzi na skutek połaczenia z aktywatorem, lub też pod wpływem związków chemicznych. Schematyczna ilustracja budowy proteasomu 26S jest przedstawiona na rysunku 1. | ||
| − | + | [[Image:Proteasom17.png|thumb|center|800px| Rysunek 1. Schemat budowy proteasomu 26S (źródło: http://atlasgeneticsoncology.org/Deep/UbiquitininCancerID20083.html).]] | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | Rysunek 1. Schemat budowy proteasomu 26S (źródło: http://atlasgeneticsoncology.org/Deep/UbiquitininCancerID20083.html). | ||
Proteasom 20S jest strukturą elastyczną a miejsca katalityczne są umiejscowione na wewnętrznej powierzchni cylindra. Tam również wiązane są substraty białkowe. Enzymatycznie aktywne są podjednostki beta1, beta2 i beta 5. Aktywność podjednostek beta jest zróżnicowana pod względem wrażliwości na inhibitory i stabilnością w pH. Trzy podstawowe aktywności enzymatyczne proteasomu obejmują: chymotrypsynopodobną (beta5), trypsynopodobną (beta 2) i kaspazopodobną (beta 1). | Proteasom 20S jest strukturą elastyczną a miejsca katalityczne są umiejscowione na wewnętrznej powierzchni cylindra. Tam również wiązane są substraty białkowe. Enzymatycznie aktywne są podjednostki beta1, beta2 i beta 5. Aktywność podjednostek beta jest zróżnicowana pod względem wrażliwości na inhibitory i stabilnością w pH. Trzy podstawowe aktywności enzymatyczne proteasomu obejmują: chymotrypsynopodobną (beta5), trypsynopodobną (beta 2) i kaspazopodobną (beta 1). | ||
Aktualna wersja na dzień 06:24, 5 kwi 2013
Proteasom
Proteasom jest złożonym kompleksem białkowym, z czego różne jego elementy mogą wykazywać odmienne właściwości enzymatyczne. Początkowo określano je mianem wielokatalitycznych kompleksów proteaz (multicatalytic proteinase complex, MPC) a od 1988 r. obowiązującą nazwą został proteasom. Proteasomy eukariotyczne występują w 2 podstawowych postaciach: 20S i 26S. Proteasom 20S nie przeprowadza proteolizy ubikwitynowanych białek komórkowych i nie ma aktywności ATP-azy. Z kolei proteasom 26S ma zdolność hydrolizy ubikwitynowanych białek a w podstawie aktywatora kompleksu zlokalizowane jest centrum aktywności ATP-azy.
Proteasom 26S (o masie 1500-2000 kDa) jest złożony z rdzenia katalitycznego 20S (700 kDa, core particie – CP) i kompleksu regulatorowego 19S (regulatory particie –RP, activator PA700). Proteasom 20S jest zbudowany z 4 pierścieni, które tworzą strukturę cylindryczną. Każdy pierścień z kolei składa się z 7 różnych podjednostek, których masa cząsteczkowa waha się pomiędzy 20 i 35 kDa. Zewnętrzne pierścienie są zbudowane z 7 podjednostek alfa (alfa 1- 7), a dwa pierścienie wewnętrzne z 7 różnych podjednostek beta (beta 1-7). Połączenie pomiędzy podjednostkami beta utrzymuje beta7. NH2-końcowe fragmenty łańcuchów podjednostek alfa zamykają kanał wiodący do wnętrza proteasomu (tzw. alfa-bramka). Otwarcie kanału i aktywacji proteasomu dochodzi na skutek połaczenia z aktywatorem, lub też pod wpływem związków chemicznych. Schematyczna ilustracja budowy proteasomu 26S jest przedstawiona na rysunku 1.
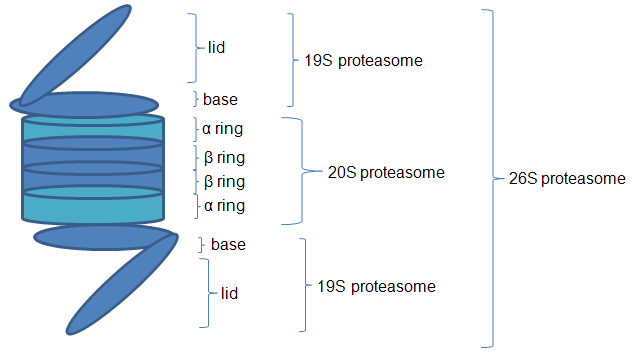
Proteasom 20S jest strukturą elastyczną a miejsca katalityczne są umiejscowione na wewnętrznej powierzchni cylindra. Tam również wiązane są substraty białkowe. Enzymatycznie aktywne są podjednostki beta1, beta2 i beta 5. Aktywność podjednostek beta jest zróżnicowana pod względem wrażliwości na inhibitory i stabilnością w pH. Trzy podstawowe aktywności enzymatyczne proteasomu obejmują: chymotrypsynopodobną (beta5), trypsynopodobną (beta 2) i kaspazopodobną (beta 1).
Proteasom 26S jest połączeniem proteasomu 20S z kompleksem aktywatora PA700 (19S). Połączone z ubikwityną białko rozpoznaje kompleks 19S a następnie otwarciu ulega kanał we wnętrzu proteasomu 20S. Kanał wejściowy bramkuje N-końcowy fragment łańcucha podjednostki alfa3. Białka wchodzące do wnętrza kanału ulegają hydrolizie w aktywnych miejscach proteolitycznych, w podjednostkach beta, na małe polipeptydy o długości do 22 aminokwasów. Cały proces zachodzi z udziałem energii z ATP.
Aktywator 19S składa się z podstawy i wieczka. Podstawę stanowi pierścień 6 ATP-az nazywanych Rpt (Rpt1-6) oraz 3 podjednostki nie posiadające aktywności ATP-azowej (Rpn1, 3 i Rpn10). Wieczko podjednostki 19S składa się wyłącznie z podjednostek Rpn (Rpn3, Rpn5-9, Rpn11-12, UCH37). Różne podjednostki spełniają odmienne funkcje w proteasomie. Rpt rozwijają i wiążą substraty białkowe. Rpn10 wiąże ubikwitynowany substrat przez domenę UIM. Rpt2 otwiera kanał proteasomu 20S oraz uczestniczy w przemieszczeniu substratu do wnętrza kanału. Rpn4 i 6 pełnią funkcje regulatorowe.
Piśmiennictwo:
- Kostur et al. Acta Haematologica Polonica 2010, 41, Nr 2, str. 261–269
- Maliński i Cichocki. Acta Haematologica Polonica 2010, 41, Nr 2, str. 261–269
- http://physrev.physiology.org/content/82/2/373.full