Kinazy JAK
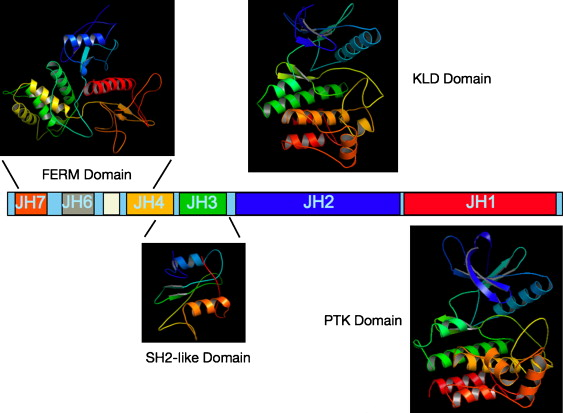
Kinazy JAK (Janus Kinase) należą do rodziny eukariotycznych niereceptorowych tyrozynowych kinaz białkowych. Ludzkie kinazy JAK1 oraz JAK2 zostały odkryte przez Wilksa w 1989 roku (Wilks 1898), który zidentyfikował ich sekwencje kodujące w oparciu o tzw. zagnieżdżony PCR. Nowe kinazy zostały wtedy nazwane Just Another Kinase. Do rodziny kinaz JAK należą również JAK3 oraz TYK2 (Tyrosine Kinase 2). Kinazy JAK są białkami wielodomenowymi o ciężarze cząsteczkowym wynoszącym około 120-140 kDa. Ich budowę ilustruje Schemat 1. Sekwencja białek JAK została podzielona na siedem regionów homologii JH1-7 (JAK Homology) z początkiem na C-końcu białek JAK (Wilks 1991). Domena katalityczna (JH1, o ciężarze cząsteczkowym około 40 kDa) znajduje się na C-końcu białka i jest funkcjonalną kinazą tyrozynową. Z domeną JH1 sąsiaduje tzw. domena pseudokinazowa JH2. Funkcja domeny JH2 polega na negatywnej regulacji aktywności katalitycznej domeny kinazowej. To właśnie te dwie sąsiadujęce ze sobą domeny zaispirowały nazwę kinaz JAK - kinazy Janusa. W starożytnej rzymskiej mitologii Janus był bożkiem o dwóch twarzach strzegącym początków i przejść, wrót i drzwi. W części N-końcowej białek JAK zostały zidentyfikowane jeszcze dwie domeny: trzecia FERM oraz czwarta SH2. Rola tych domen polega przede wszystkim na asocjacji i kotwiczeniu całego białka do cytoplazmatycznej cześci receptorów cytokin, czynników wzrostu oraz hormonów.
| kinaza JAK | para tyrozyn |
|---|---|
| JAK1 | Tyr1038/Tyr1039 |
| JAK2 | Tyr1007/Tyr1008 |
| JAK3 | Tyr980/Tyr981 |
| TYK2 | Tyr1054/Tyr1055 |
Ważną częścią struktury kinaz JAK jest pętla aktywacyjna, w której zlokalizowane są chrakterystyczne dla kinaz JAK ewolucyjnie konserwowane pary tyrozyn (przedstawione w Tabeli 1). Mechanizm aktywacji kinaz JAK opiera się na autofosforylacji tych reszt tyrozyny. Dołączenie rszt fosforanów powoduje otwarcie pętli aktywacyjnej i przesunięcie motywu DFG do konformacji aktywnej DFG-in, zdolnego do wiązania kationów magnezu. Autofosforylacja umożliwia również wiązanie wielu białek regulatorowych, np.: SOCS, PTP. Charakterystyczna dla kinaz JAK jest również tzw. pętla insercyjna również zaangażowana w regulację aktywności fosforylacyjnej tych kinaz (Lucet 2006).
Ścieżka JAK/STAT
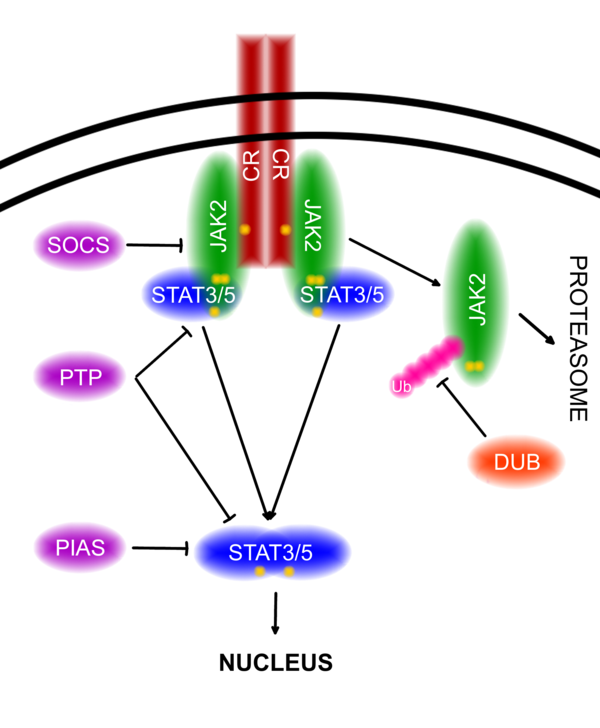
Kinazy JAK są częścią sieci sygnalnej JAK/STAT (Signal Transducer and Activator of Transcription), które przewodzą sygnały zewnątrzkomórkowe pochodzące od cytokin, interferonów czy czynników wzrostu. Wiązanie liganda do receptora transmembranowego prowadzi do jego dimeryzacji i aktywacji, a w konsekwencji do autofosforylacji i atywacji kinaz JAK (Rawlings et al. 2004). Białka STAT kotwiczą do fosforylowanego receptora za pośrednictwem domen SH2 (Yamaoka et al. 2004). Fosforylacja białek STAT przez kinazy JAK umożliwia ich dimeryzacji oraz translokacji do jądra komórkowego. Najczęściej pojawiające się heterodimery białaek STAT to: STAT1:STAT2, STAT1:STAT3 oraz STAT5A:STAT5B. Proces translokacji jest zależny od importyny a-5, a także od ścieżki jądrowego importu zależnej od GTP-azy Ran (Rawlings et al. 2004).
Najważniejsze białka efektorowe o roli regulatorowej ścieżki JAK/STAT, zilustrowane na Schemacie 2 to (Greenhalgh & Hilton 2001, Rawlings 2004):
- PIAS (protein inhibitor of activated STAT), wiążą dimery białek STAT przecidziałając ich oddziąływaniu z odpowiednimi regionami promotorowymi DNA (Rawlings 2004);
- PTP (protein tyrosine phosphatase) takie, jak: SHP-1, SHP-2 (SH2 containing phosphatase) mogą defosforylować kinazy JAK oraz receptory (Valentino and Pierre 2006, Schindler and Plumlee2008);
- SH2/Lnk/APS (Src homology 2 / Linker of T-cell receptor pathways / prostate specific antigen (kallikrein-related peptidase)) zawierają domenę homologiczną do plekstryny, jak również domenę SH2 i moga być substratami kinaz JAK (Rawlings 2004);
- SOCS (suppressor of cytokine signaling) regulują ścieżkę JAK/STAT na zasadzie sprzężenia zwrotnego: białka STAT indukują ekspresję genów socs, których produkty wiążą sie z fosforylowanymi kinazami JAK oraz ich receptorami, przeciwdziałając transdukcji sygnału (Valentino and Pierre 2006)
- STAM (signal-transducing adapter molecule) ułatwiają aktywację transkrypcyjną niektórych genów, np. myc (Lohi & Lehto 2001)
- StIP (STAT-interacting protein, WD40 protein) służą jako białka adaptorowe - zdolne do asocjacji zarówno z kiianazami JAK, jak i nieufosforylowanymi białkami STAT (O'Shea et al. 2002).