(→Kinazy JAK) |
(→Kinazy JAK) |
||
| Linia 1: | Linia 1: | ||
==Kinazy JAK== | ==Kinazy JAK== | ||
| − | Kinazy JAK (<u>Ja</u>nus <u>K</u>inase) należą do rodziny eukariotycznych niereceptorowych tyrozynowych [[Kinazy białkowe|kinaz białkowych]]. Ludzkie kinazy [[JAK1]] oraz [[JAK2]] zostały odkryte przez Wilksa w 1989 roku | + | Kinazy JAK (<u>Ja</u>nus <u>K</u>inase) należą do rodziny eukariotycznych niereceptorowych tyrozynowych [[Kinazy białkowe|kinaz białkowych]]. Ludzkie kinazy [[JAK1]] oraz [[JAK2]] zostały odkryte przez Wilksa w 1989 roku [1], który zidentyfikował ich sekwencje kodujące w oparciu o tzw. zagnieżdżony PCR. Te nowe wtedy kinazy zostały nazwane <u>J</u>ust <u>A</u>nother <u>K</u>inase. Do rodziny kinaz JAK należą ponadto [[JAK3]] oraz [[TYK2]] (<u>Ty</u>rosine <u>K</u>inase 2). |
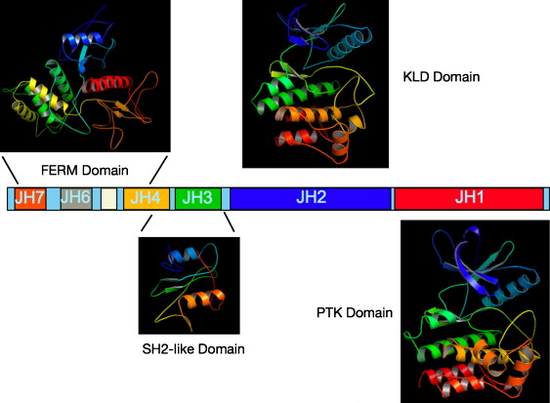
| − | Kinazy JAK są białkami wielodomenowymi o ciężarze cząsteczkowym wynoszącym około 120-140 kDa. Ich budowę ilustruje Schemat 1. Sekwencja białek JAK została podzielona na siedem regionów homologii JH1-7 (<u>J</u>AK <u>H</u>omology) z początkiem na C-końcu | + | Kinazy JAK są białkami wielodomenowymi o ciężarze cząsteczkowym wynoszącym około 120-140 kDa. Ich budowę ilustruje Schemat 1. Sekwencja białek JAK została podzielona na siedem regionów homologii JH1-7 (<u>J</u>AK <u>H</u>omology) z początkiem na C-końcu [2]. |
[[Image: JAK-Rysunek1.png|thumb|center|550px|Schemat 1: Struktura domenowa kinaz JAK, PTK - Protein Tyrosine Kinase, KLD - Kinase-like Domain (żródło: A.F.Wilks ''Seminars in Cell & Developmental Biology'' 19(2008): 319-328)]] | [[Image: JAK-Rysunek1.png|thumb|center|550px|Schemat 1: Struktura domenowa kinaz JAK, PTK - Protein Tyrosine Kinase, KLD - Kinase-like Domain (żródło: A.F.Wilks ''Seminars in Cell & Developmental Biology'' 19(2008): 319-328)]] | ||
| − | Domena katalityczna ('''JH1''', o ciężarze cząsteczkowym około 40 kDa) znajduje się na C-końcu białka i jest funkcjonalną kinazą tyrozynową. Z domeną JH1 sąsiaduje tzw. domena pseudokinazowa '''JH2'''. To właśnie te dwie sąsiadujęce ze sobą domeny zainspirowały nazwę kinaz JAK - kinazy Janusa. W mitologii starożytnego Rzymu Janus był bożkiem o dwóch twarzach strzegącym początków i przejść, wrót i drzwi. Funkcja domeny JH2 (KLD - Kinase-like Domain) polega na negatywnej regulacji aktywności katalitycznej domeny kinazowej JH1. Pomimo homologii do domeny kinazowej, jest ona pozbawiona kluczowych aminokwasów katalitycznych, a tym samym aktywności kinazowej. Na przykład motyw DFG został zastąpiony przez DPG | + | Domena katalityczna ('''JH1''', o ciężarze cząsteczkowym około 40 kDa) znajduje się na C-końcu białka i jest funkcjonalną kinazą tyrozynową. Z domeną JH1 sąsiaduje tzw. domena pseudokinazowa '''JH2'''. To właśnie te dwie sąsiadujęce ze sobą domeny zainspirowały nazwę kinaz JAK - kinazy Janusa. W mitologii starożytnego Rzymu Janus był bożkiem o dwóch twarzach strzegącym początków i przejść, wrót i drzwi. Funkcja domeny JH2 (KLD - Kinase-like Domain) polega na negatywnej regulacji aktywności katalitycznej domeny kinazowej JH1. Pomimo homologii do domeny kinazowej, jest ona pozbawiona kluczowych aminokwasów katalitycznych, a tym samym aktywności kinazowej. Na przykład motyw DFG został zastąpiony przez DPG [3]. Usunięcie domeny JH2 prowadzi do konstytutywnej aktywacji kinaz JAK [4,5]. |
W części N-końcowej białek JAK zostały zidentyfikowane dwie dodatkowe domeny (oprócz JH1 i JH2): '''FERM''' oraz '''SH2'''. Rola tych domen polega przede wszystkim na asocjacji całego białka do cytoplazmatycznej cześci receptorów cytokin, czynników wzrostu oraz hormonów. Domena SH2 (Src Homology) występuje u wielu cytoplazmatycznych kinaz tyrozynowych. Obejmuje region JH3 oraz częściowo JH4. Uczestniczy w oddziaływaniu z fosfotyrozynami białek sygnałowych. Domena FERM (Four ponit one, Ezrin, Radixin, Moesin Homology) znajduje się na N-końcu białek JAK (regiony JH4 do JH7). Zbudowana jest z trzech subdomen: F1 homologicznej do ubikwityny, F2 - do białek wiążących acylo-CoA, a także F3 - do plekstryny. Jej główna rola to asocjacja z receptorami. | W części N-końcowej białek JAK zostały zidentyfikowane dwie dodatkowe domeny (oprócz JH1 i JH2): '''FERM''' oraz '''SH2'''. Rola tych domen polega przede wszystkim na asocjacji całego białka do cytoplazmatycznej cześci receptorów cytokin, czynników wzrostu oraz hormonów. Domena SH2 (Src Homology) występuje u wielu cytoplazmatycznych kinaz tyrozynowych. Obejmuje region JH3 oraz częściowo JH4. Uczestniczy w oddziaływaniu z fosfotyrozynami białek sygnałowych. Domena FERM (Four ponit one, Ezrin, Radixin, Moesin Homology) znajduje się na N-końcu białek JAK (regiony JH4 do JH7). Zbudowana jest z trzech subdomen: F1 homologicznej do ubikwityny, F2 - do białek wiążących acylo-CoA, a także F3 - do plekstryny. Jej główna rola to asocjacja z receptorami. | ||
| Linia 28: | Linia 28: | ||
|} | |} | ||
| − | Ważną częścią struktury kinaz JAK jest pętla aktywacyjna, w której zlokalizowane są charakterystyczne dla kinaz JAK ewolucyjnie konserwowane '''pary tyrozyn''' (przedstawione w Tabeli 1). Mechanizm aktywacji kinaz JAK opiera się na autofosforylacji tych reszt tyrozyny. Dołączenie reszt fosforanów powoduje otwarcie pętli aktywacyjnej i przesunięcie motywu DFG do konformacji aktywnej ''DFG-in'', zdolnego do wiązania kationów magnezu. Autofosforylacja umożliwia również wiązanie wielu białek regulatorowych, np.: SOCS, PTP. Charakterystyczna dla kinaz JAK jest również tzw. pętla insercyjna zaangażowana w regulację ich aktywności fosforylacyjnej | + | Ważną częścią struktury domeny kinazowej kinaz JAK jest pętla aktywacyjna, w której zlokalizowane są charakterystyczne dla kinaz JAK ewolucyjnie konserwowane '''pary tyrozyn''' (przedstawione w Tabeli 1). Mechanizm aktywacji kinaz JAK opiera się na autofosforylacji tych reszt tyrozyny. Dołączenie reszt fosforanów powoduje otwarcie pętli aktywacyjnej i przesunięcie motywu DFG do konformacji aktywnej ''DFG-in'', zdolnego do wiązania kationów magnezu. Autofosforylacja umożliwia również wiązanie wielu białek regulatorowych, np.: SOCS, PTP. Charakterystyczna dla kinaz JAK jest również tzw. pętla insercyjna zaangażowana w regulację ich aktywności fosforylacyjnej [6]. |
'''Ścieżka JAK/STAT''' | '''Ścieżka JAK/STAT''' | ||
---- | ---- | ||
| − | Kinazy JAK są częścią sieci sygnalnej JAK/STAT (<u>S</u>ignal <u>T</u>ransducer and <u>A</u>ctivator of <u>T</u>ranscription), która przewodzi sygnały zewnątrzkomórkowe pochodzące od cytokin, interferonów oraz czynników wzrostu. Wiązanie liganda do receptora transmembranowego prowadzi do jego dimeryzacji i aktywacji, a w konsekwencji do autofosforylacji i atywacji kinaz JAK | + | Kinazy JAK są częścią sieci sygnalnej JAK/STAT (<u>S</u>ignal <u>T</u>ransducer and <u>A</u>ctivator of <u>T</u>ranscription), która przewodzi sygnały zewnątrzkomórkowe pochodzące od cytokin, interferonów oraz czynników wzrostu. Wiązanie liganda do receptora transmembranowego prowadzi do jego dimeryzacji i aktywacji, a w konsekwencji do autofosforylacji i atywacji kinaz JAK [7]. Białka STAT kotwiczą do ufosforylowanego receptora za pośrednictwem domen SH2 [8]. Fosforylacja białek STAT przez kinazy JAK umożliwia ich dimeryzację oraz translokację do jądra komórkowego. Heterodimery białek STAT pojawiające się najczęściej to: STAT1:STAT2, STAT1:STAT3 oraz STAT5A:STAT5B. Proces translokacji czynników transkrypcyjnych STAT do jądra komórkowego jest zależny od importyny a-5, a także od jądrowego importu zależnego od GTP-azy Ran [7]. |
[[Image: JAK-Rysunek2.png|thumb|right|350px|Schemat 2: Elementy negatywnej regulacji ścieżek JAK/STAT (Ub - ubiquitin, DUB - deubiquitinase)]] | [[Image: JAK-Rysunek2.png|thumb|right|350px|Schemat 2: Elementy negatywnej regulacji ścieżek JAK/STAT (Ub - ubiquitin, DUB - deubiquitinase)]] | ||
| − | Najważniejsze białka efektorowe o roli regulatorowej ścieżek JAK/STAT, zilustrowane na Schemacie 2, to | + | Najważniejsze białka efektorowe o roli regulatorowej ścieżek JAK/STAT, zilustrowane na Schemacie 2, to: |
| − | * PIAS (Protein Inhibitor of Activated STAT), wiążą dimery białek STAT przeciwdziałając ich wiązaniu do odpowiednich regionów promotorowych DNA | + | * PIAS (Protein Inhibitor of Activated STAT), wiążą dimery białek STAT przeciwdziałając ich wiązaniu do odpowiednich regionów promotorowych DNA [7,9]; |
| − | * PTP (Protein Tyrosine Phosphatase) takie, jak: SHP-1, SHP-2 (SH2 Containing Phosphatase) mogą defosforylować kinazy JAK oraz receptory | + | * PTP (Protein Tyrosine Phosphatase) takie, jak: SHP-1, SHP-2 (SH2 Containing Phosphatase) mogą defosforylować kinazy JAK oraz receptory [10,11]; |
| − | + | * SOCS (Suppressor of Cytokine Signaling), regulują ścieżkę JAK/STAT na zasadzie sprzężenia zwrotnego: białka STAT indukują ekspresję genów socs, których produkty wiążą sie z fosforylowanymi kinazami JAK oraz ich receptorami, przeciwdziałając transdukcji sygnału [9,10]; | |
| − | * SOCS (Suppressor of Cytokine Signaling), regulują ścieżkę JAK/STAT na zasadzie sprzężenia zwrotnego: białka STAT indukują ekspresję genów socs, których produkty wiążą sie z fosforylowanymi kinazami JAK oraz ich receptorami, przeciwdziałając transdukcji sygnału | + | * STAM (Signal Transducing Adapter Molecule), ułatwiają aktywację transkrypcyjną niektórych genów, np. myc [12]; |
| − | * STAM (Signal Transducing Adapter Molecule), ułatwiają aktywację transkrypcyjną niektórych genów, np. myc | + | * STIP (STAT-interacting Protein, WD40 protein), służą jako białka adaptorowe - zdolne do asocjacji zarówno z kinazami JAK, jak i nieufosforylowanymi białkami STAT [13]; |
| − | * STIP (STAT-interacting Protein, WD40 protein), służą jako białka adaptorowe - zdolne do asocjacji zarówno z kinazami JAK, jak i nieufosforylowanymi białkami STAT (O'Shea | + | * SH2/Lnk/APS (Src Homology 2 / Linker of T-cell Receptor Pathways / Prostate Specific Antigen (kallikrein-related peptidase)), zawierają domenę homologiczną do plekstryny, jak również domenę SH2 i moga być substratami kinaz JAK [7]. |
| + | |||
| + | '''Literatura''' | ||
| + | ---- | ||
| + | # Wilks, A.F. Two putative protein-tyrosine kinases identified by application of the polymerase chain reaction. Proceedings of the National Academy of Sciences, 1989, 86 (5), 1603-1607. | ||
| + | # Wilks, A.F.; Harpur, A.G.; Kurban, R.R.; Ralph, S.J.; Zürcher, G.; Ziemiecki, A. Two novel protein-tyrosine kinases, each with a second phosphotransferase-related catalytic domain, define a new class of protein kinase. Mol. Cell. Biol., 1991, 11 (4), 2057-2065. | ||
| + | # Wilks, A.F. The JAK kinases: Not just another kinase drug discovery target. Seminars in Cell & Developmental Biology, 2008, 19 (4), 319-328. | ||
| + | # Saharinen, P.; Silvennoinen, O. The Pseudokinase Domain Is Required for Suppression of Basal Activity of Jak2 and Jak3 Tyrosine Kinases and for Cytokine-inducible Activation of Signal Transduction. J. Biol. Chem., 2002, 277 (49), 47954-47963. | ||
| + | # Saharinen, P.; Vihinen, M.; Silvennoinen, O. Autoinhibition of Jak2 Tyrosine Kinase Is Dependent on Specific Regions in Its Pseudokinase Domain. Mol. Biol. Cell, 2003, 14 (4), 1448-1459. | ||
| + | # Lucet, I.S.; Fantino, E.; Styles, M.; Bamert, R.; Patel, O.; Broughton, S.E.; Walter, M.; Burns, C.J.; Treutlein, H.; Wilks, A.F.; Rossjohn, J. The structural basis of Janus kinase 2 inhibition by a potent and specific pan-Janus kinase inhibitor. Blood, 2006, 107 (1), 176-183. | ||
| + | # Rawlings, J.S.; Rosler, K.M.; Harrison, D.A. The JAK/STAT signaling pathway. J. Cell Sci., 2004, 117 (8), 1281-1283. | ||
| + | # Yamaoka, K.; Saharinen, P.; Pesu, M.; Holt, V.; Silvennoinen, O.; O'Shea, J. The Janus kinases (Jaks). Genome Biology, 2004, 5 (12), 253. | ||
| + | # Greenhalgh, C.J.; Hilton, D.J. Negative regulation of cytokine signaling. J. Leukoc. Biol., 2001, 70 (3), 348-356. | ||
| + | # Valentino, L.; Pierre, J. JAK/STAT signal transduction: Regulators and implication in hematological malignancies. Biochem. Pharmacol., 2006, 71 (6), 713-721. | ||
| + | # Schindler, C.; Plumlee, C. Inteferons pen the JAK/STAT pathway. Seminars in Cell & Developmental Biology, 2008, 19 (4), 311-318. | ||
| + | # Lohi, O.; Lehto, V.-P. STAM/EAST/Hbp adapter proteins - integrators of signalling pathways. FEBS Lett., 2001, 508 (3), 287-290. | ||
| + | # O'Shea, J.J.; Gadina, M.; Schreiber, R.D. Cytokine Signaling in 2002: New Surprises in the Jak/Stat Pathway. Cell, 2002, 109 (2), S121-S131. | ||
Wersja z 09:31, 12 maj 2014
Kinazy JAK
Kinazy JAK (Janus Kinase) należą do rodziny eukariotycznych niereceptorowych tyrozynowych kinaz białkowych. Ludzkie kinazy JAK1 oraz JAK2 zostały odkryte przez Wilksa w 1989 roku [1], który zidentyfikował ich sekwencje kodujące w oparciu o tzw. zagnieżdżony PCR. Te nowe wtedy kinazy zostały nazwane Just Another Kinase. Do rodziny kinaz JAK należą ponadto JAK3 oraz TYK2 (Tyrosine Kinase 2).
Kinazy JAK są białkami wielodomenowymi o ciężarze cząsteczkowym wynoszącym około 120-140 kDa. Ich budowę ilustruje Schemat 1. Sekwencja białek JAK została podzielona na siedem regionów homologii JH1-7 (JAK Homology) z początkiem na C-końcu [2].
Domena katalityczna (JH1, o ciężarze cząsteczkowym około 40 kDa) znajduje się na C-końcu białka i jest funkcjonalną kinazą tyrozynową. Z domeną JH1 sąsiaduje tzw. domena pseudokinazowa JH2. To właśnie te dwie sąsiadujęce ze sobą domeny zainspirowały nazwę kinaz JAK - kinazy Janusa. W mitologii starożytnego Rzymu Janus był bożkiem o dwóch twarzach strzegącym początków i przejść, wrót i drzwi. Funkcja domeny JH2 (KLD - Kinase-like Domain) polega na negatywnej regulacji aktywności katalitycznej domeny kinazowej JH1. Pomimo homologii do domeny kinazowej, jest ona pozbawiona kluczowych aminokwasów katalitycznych, a tym samym aktywności kinazowej. Na przykład motyw DFG został zastąpiony przez DPG [3]. Usunięcie domeny JH2 prowadzi do konstytutywnej aktywacji kinaz JAK [4,5].
W części N-końcowej białek JAK zostały zidentyfikowane dwie dodatkowe domeny (oprócz JH1 i JH2): FERM oraz SH2. Rola tych domen polega przede wszystkim na asocjacji całego białka do cytoplazmatycznej cześci receptorów cytokin, czynników wzrostu oraz hormonów. Domena SH2 (Src Homology) występuje u wielu cytoplazmatycznych kinaz tyrozynowych. Obejmuje region JH3 oraz częściowo JH4. Uczestniczy w oddziaływaniu z fosfotyrozynami białek sygnałowych. Domena FERM (Four ponit one, Ezrin, Radixin, Moesin Homology) znajduje się na N-końcu białek JAK (regiony JH4 do JH7). Zbudowana jest z trzech subdomen: F1 homologicznej do ubikwityny, F2 - do białek wiążących acylo-CoA, a także F3 - do plekstryny. Jej główna rola to asocjacja z receptorami.
| kinaza JAK | para tyrozyn |
|---|---|
| JAK1 | Tyr1038/Tyr1039 |
| JAK2 | Tyr1007/Tyr1008 |
| JAK3 | Tyr980/Tyr981 |
| TYK2 | Tyr1054/Tyr1055 |
Ważną częścią struktury domeny kinazowej kinaz JAK jest pętla aktywacyjna, w której zlokalizowane są charakterystyczne dla kinaz JAK ewolucyjnie konserwowane pary tyrozyn (przedstawione w Tabeli 1). Mechanizm aktywacji kinaz JAK opiera się na autofosforylacji tych reszt tyrozyny. Dołączenie reszt fosforanów powoduje otwarcie pętli aktywacyjnej i przesunięcie motywu DFG do konformacji aktywnej DFG-in, zdolnego do wiązania kationów magnezu. Autofosforylacja umożliwia również wiązanie wielu białek regulatorowych, np.: SOCS, PTP. Charakterystyczna dla kinaz JAK jest również tzw. pętla insercyjna zaangażowana w regulację ich aktywności fosforylacyjnej [6].
Ścieżka JAK/STAT
Kinazy JAK są częścią sieci sygnalnej JAK/STAT (Signal Transducer and Activator of Transcription), która przewodzi sygnały zewnątrzkomórkowe pochodzące od cytokin, interferonów oraz czynników wzrostu. Wiązanie liganda do receptora transmembranowego prowadzi do jego dimeryzacji i aktywacji, a w konsekwencji do autofosforylacji i atywacji kinaz JAK [7]. Białka STAT kotwiczą do ufosforylowanego receptora za pośrednictwem domen SH2 [8]. Fosforylacja białek STAT przez kinazy JAK umożliwia ich dimeryzację oraz translokację do jądra komórkowego. Heterodimery białek STAT pojawiające się najczęściej to: STAT1:STAT2, STAT1:STAT3 oraz STAT5A:STAT5B. Proces translokacji czynników transkrypcyjnych STAT do jądra komórkowego jest zależny od importyny a-5, a także od jądrowego importu zależnego od GTP-azy Ran [7].
Najważniejsze białka efektorowe o roli regulatorowej ścieżek JAK/STAT, zilustrowane na Schemacie 2, to:
- PIAS (Protein Inhibitor of Activated STAT), wiążą dimery białek STAT przeciwdziałając ich wiązaniu do odpowiednich regionów promotorowych DNA [7,9];
- PTP (Protein Tyrosine Phosphatase) takie, jak: SHP-1, SHP-2 (SH2 Containing Phosphatase) mogą defosforylować kinazy JAK oraz receptory [10,11];
- SOCS (Suppressor of Cytokine Signaling), regulują ścieżkę JAK/STAT na zasadzie sprzężenia zwrotnego: białka STAT indukują ekspresję genów socs, których produkty wiążą sie z fosforylowanymi kinazami JAK oraz ich receptorami, przeciwdziałając transdukcji sygnału [9,10];
- STAM (Signal Transducing Adapter Molecule), ułatwiają aktywację transkrypcyjną niektórych genów, np. myc [12];
- STIP (STAT-interacting Protein, WD40 protein), służą jako białka adaptorowe - zdolne do asocjacji zarówno z kinazami JAK, jak i nieufosforylowanymi białkami STAT [13];
- SH2/Lnk/APS (Src Homology 2 / Linker of T-cell Receptor Pathways / Prostate Specific Antigen (kallikrein-related peptidase)), zawierają domenę homologiczną do plekstryny, jak również domenę SH2 i moga być substratami kinaz JAK [7].
Literatura
- Wilks, A.F. Two putative protein-tyrosine kinases identified by application of the polymerase chain reaction. Proceedings of the National Academy of Sciences, 1989, 86 (5), 1603-1607.
- Wilks, A.F.; Harpur, A.G.; Kurban, R.R.; Ralph, S.J.; Zürcher, G.; Ziemiecki, A. Two novel protein-tyrosine kinases, each with a second phosphotransferase-related catalytic domain, define a new class of protein kinase. Mol. Cell. Biol., 1991, 11 (4), 2057-2065.
- Wilks, A.F. The JAK kinases: Not just another kinase drug discovery target. Seminars in Cell & Developmental Biology, 2008, 19 (4), 319-328.
- Saharinen, P.; Silvennoinen, O. The Pseudokinase Domain Is Required for Suppression of Basal Activity of Jak2 and Jak3 Tyrosine Kinases and for Cytokine-inducible Activation of Signal Transduction. J. Biol. Chem., 2002, 277 (49), 47954-47963.
- Saharinen, P.; Vihinen, M.; Silvennoinen, O. Autoinhibition of Jak2 Tyrosine Kinase Is Dependent on Specific Regions in Its Pseudokinase Domain. Mol. Biol. Cell, 2003, 14 (4), 1448-1459.
- Lucet, I.S.; Fantino, E.; Styles, M.; Bamert, R.; Patel, O.; Broughton, S.E.; Walter, M.; Burns, C.J.; Treutlein, H.; Wilks, A.F.; Rossjohn, J. The structural basis of Janus kinase 2 inhibition by a potent and specific pan-Janus kinase inhibitor. Blood, 2006, 107 (1), 176-183.
- Rawlings, J.S.; Rosler, K.M.; Harrison, D.A. The JAK/STAT signaling pathway. J. Cell Sci., 2004, 117 (8), 1281-1283.
- Yamaoka, K.; Saharinen, P.; Pesu, M.; Holt, V.; Silvennoinen, O.; O'Shea, J. The Janus kinases (Jaks). Genome Biology, 2004, 5 (12), 253.
- Greenhalgh, C.J.; Hilton, D.J. Negative regulation of cytokine signaling. J. Leukoc. Biol., 2001, 70 (3), 348-356.
- Valentino, L.; Pierre, J. JAK/STAT signal transduction: Regulators and implication in hematological malignancies. Biochem. Pharmacol., 2006, 71 (6), 713-721.
- Schindler, C.; Plumlee, C. Inteferons pen the JAK/STAT pathway. Seminars in Cell & Developmental Biology, 2008, 19 (4), 311-318.
- Lohi, O.; Lehto, V.-P. STAM/EAST/Hbp adapter proteins - integrators of signalling pathways. FEBS Lett., 2001, 508 (3), 287-290.
- O'Shea, J.J.; Gadina, M.; Schreiber, R.D. Cytokine Signaling in 2002: New Surprises in the Jak/Stat Pathway. Cell, 2002, 109 (2), S121-S131.