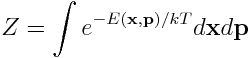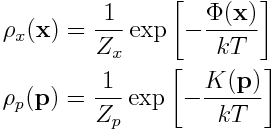(→Energia swobodna) |
(→Energia swobodna) |
||
| Linia 64: | Linia 64: | ||
==Potencjał średnich sił== | ==Potencjał średnich sił== | ||
Znaczna część metod wyznaczania energii swobodnej koncentruje się na wyznaczeniu tzw. potencjału średnich sił (Potential of Mean Force, '''PMF'''). | Znaczna część metod wyznaczania energii swobodnej koncentruje się na wyznaczeniu tzw. potencjału średnich sił (Potential of Mean Force, '''PMF'''). | ||
| + | [[Image:Angle-140_dihedral_2000x2000.png |thumb|right]] | ||
| + | [[Image:ProbDensity.png |thumb|right]] | ||
| + | [[Image:Pmf.png |thumb|right]] | ||
| + | PMF jest funkcją parametru (zwykle oznaczanego jako ξ), który określa przebieg reakcji (wiązania ligandu, przejścia cząsteczki przez błonę, zmiany konformacyjnej, itd.). | ||
| + | Energię swobodną | ||
Wersja z 18:48, 9 lut 2015
Spis treści
Energia swobodna
Energia swobodna jest wielkością fizyczną przydatną w opisie układów, będących w kontakcie z otoczeniem (tzn. oddziaływujących z nim). Pojęcie to wywodzi się z termodynamiki fenomenologicznej, gdzie służyło m.in. do określenia maksymalnej pracy, jaką można uzyskać ze zgromadzonej w układzie energii (stąd określenie swobodna), przy określonym typie oddziaływań z otoczeniem (termiczne, mechaniczne, materialne). Jednakże energia swobodna (i jej pochodne cząstkowe) pozwala wyznaczyć wszystkie makroskopowe równowagowe własności układu.
Zarys historyczny
W zestawie artykułów Energia swobodna, które znaleźć można w ramach tego wiki, ograniczamy sie do opisu energii swobodnej Helmholtza:
czyli potencjału termodynamicznego stosowanego w przypadku układów w kontakcie termicznym z otoczeniem (przy stałej liczbie cząsteczek, N, i stałej objętości, V). Energia swobodna Helmholtza w powyższej postaci ograniczona jest do układów makroskopowych, tzn. zbudowanych z niewyobrażalnie dużej liczby cząsteczek. Należy podkreślić, że termodynamika fenomenologiczna nie zakłada, że otaczająca nas materia zbudowana jest z atomów, a jedynie postuluje istnienie i właściwości dwóch funkcji: energii wewnętrznej (U) i entropii (S).
Postać mikroskopowa energii swobodnej
Ponieważ zajmujemy się układami w skali mikro (białka, kwasy nukleinowe itd.), interesować nas będzie postać energii swobodnej Helmholtza, wynikająca z postulatów mechaniki statystycznej. To znaczy,
gdzie w miejsce energii wewnętrzenej pojawiła się wartość oczekiwana energii całkowitej, E, przy gęstości prawdopodobieństwa:
zwanej rozkładem Boltzmanna. Stała k występująca w powyższych równaniach nosi nazwę stałej Boltzmanna, natomiast stała Z jest stałą normalizacyjną:
Ludwig Boltzmann, twórca jednego z najważniejszych działów fizyki - mechaniki statystycznej - zasłużył się, proponując następującą postać funkcyjną entropii:
Zauważmy, że do wyznaczenia energii swobodnej potrzebna jest znajomość energii całkowitej układu, która jest funkcją położeń, x, i pędów atomów, p, z których zbudowany jest układ.
Dlaczego energia swobodna jest taka ważna?
Energia swobodna ma bardzo prostą interpretację probabilistyczną, która bezpośrednio łączy się z naszymi intuicjami związanymi z wieloma zjawiskami molekularnymi (wiązanie ligandów przez enzymy, praca pomp transmembranowych, asocjacja białek itd.). Jednakże aby do tej "prostej interpretacji probabilistycznej" dojść, potrzebujemy zaobserwować kilka własności energii swobodnej. Dokładne wyprowadzenia znaleźć można w [1,2].
1. Rozkład prawdopodobieństwa położeń, x, jest niezależny od rozkładu prowdopodobieństwa pędów, p. To znaczy, rozkład Boltzmanna zapisać można jako:
gdzie
Grecka litera Φ oznacza energię potencjalną układu (w funkcji położeń), zaś K to energia kinetyczna.
2. Po przekształceniu, mikroskopowe równanie na energię swobodną można zapisać w postaci:
Całkowanie przebiega po całej przestrzeni konfiguracyjnej (tzn. po wszystkich możliwych położeniach atomów x). Większość konfiguracji (zwłaszcza tych, które odznaczają się niekorzystnymi zawadami sterycznymi) będzie miało bardzo wysoką energię potencjalną, stąd ich wkład do energii swobodnej będzie pomijalny. Zapiszmy powyższe równanie w nieco innej postaci:
Zauważmy teraz, że jeśli interesuje nas jakiś konkretny stan, A, naszego układu - przykładowo, enzym związany z ligandem - to jest on w istocie pewnym podzbiorem przestrzeni konfiguracyjnej. Możemy więc prawdopodobieństwo znalezienia układu w stanie A wyrazić za pomocą całki:
Powyższe wyrażenie przypomina poprzednie wyrażenie na energię swobodną, w którym brakuje stałej normalizacyjnej, oraz obszaru całkowania A.
Nasuwa się pomysł, aby zdefiniować energię swobodną dla stanu A jako:
skąd
Oznaczmy jako B jakiś inny stan układu (np. enzym niezwiązany z ligandem). Wówczas stosunek prawdopodobieństw A i B powiązać można z różnicą energii swobodnych:
Potencjał średnich sił
Znaczna część metod wyznaczania energii swobodnej koncentruje się na wyznaczeniu tzw. potencjału średnich sił (Potential of Mean Force, PMF).
PMF jest funkcją parametru (zwykle oznaczanego jako ξ), który określa przebieg reakcji (wiązania ligandu, przejścia cząsteczki przez błonę, zmiany konformacyjnej, itd.). Energię swobodną
Literatura
- Molecular Modelling: Principles and Applications (2nd Edition), A. Leach.
- Understanding Molecular Simulation (2nd Edition): From Algorithms to Applications, D. Frenkel.