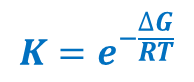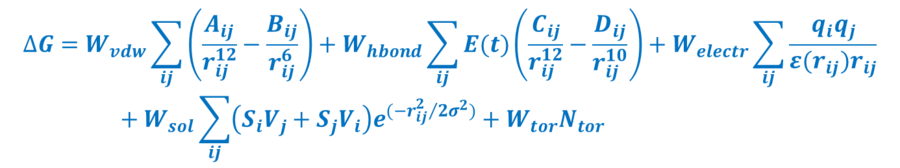Dokowanie molekularne
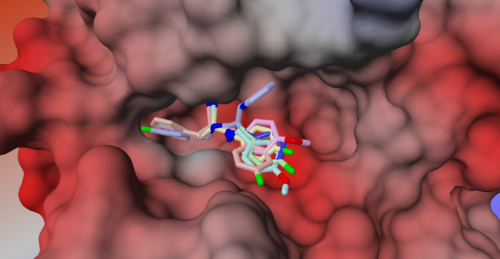
Dokowanie molekularne jest dziedziną modelowania molekularnego, która polega na poszukiwaniu optymalnego modu wiązania liganda w strukturze receptora (oddziaływania liganda z receptorem). Zbiór wszystkich konfiguracji liganda i receptora tworzy przestrzeń konformacyjną z charakterystycznym dla siebie układem powierzchni energii potencjalnej (w przyjętym polu siłowym). Dokowanie molekularne poszukuje minimum globalnego pola siłowego wyznaczonego dla kompleksu ligand-receptor.
Realizacja tego celu wymaga opracowania dwóch głównych metod: algorytmu przeszukiwania wielowymiarowej przestrzeni konfiguracyjnej, a także funkcji oceniającej (scoring function) zaproponowane konfiguracje układu ligand-receptor.
Dokowanie molekularne może być pomocne w projektowaniu nowych ligandów działających jak inhibitory (potencjalne leki). Z drugiej strony dokowanie może być pomocne w badaniu mechanizmu działania znanych inhibitorów, np. określaniu miejsca i sposobu oddziaływania ze znanym białkowym celem molekularnym (hamowanym enzymem).
Wielkość przestrzeni konfiguracyjnej, a więc możliwych stanów, jakie może przyjąć układ liganda oddziałującego z receptorem (np. białko, kwas nukleinowy) rośnie wykładniczo wraz z liczbą wymiarów D (liczbą stopni swobody układu: liganda i receptora). Jeśli przyjmiemy taki sam stopień skomplikowania - możliwych wartości N dla każdego z wymiarów, to wielkość przestrzeni konfiguracyjnej (konformacyjnej) będzie równa
Wymiarami mogą być np.: położenie liganda, orientacja cząsteczki liganda czy swobodne wiązanie w cząsteczce liganda (z możliwością rotacji). Pełna swoboda konformacyjna receptora powoduje znaczący wzrost wielkości przestrzeni konformacyjnej ze względu na wielkość jego cząsteczki. Często zatem ogranicza się pełną swobodę konformacyjną do cząsteczki liganda w dokowaniu tzw. rigid-flexible.
Ze względu na znaczny koszt obliczeniowy systematycznego i wyczerpującego przeszukania przestrzeni konfiguracyjnej, algorytmy przeszukania opracowane dla potrzeb dokowania molekularnego to najczęściej algorytmy heurystyczne i stochastyczne. Algorytmy te próbkują przestrzeń konfiguracyjną w sposób losowy (w przeciwieństwie do algorytmów deterministycznych i systematycznych). W związku z tym proponują one przybliżone rozwiązanie problemu, które nie musi być rozwiązaniem globalnym, optymalnym. Wynik dokowania nie musi być optymalny. Przykładowe algorytmy przeszukiwania to: symulowane wyżarzanie typu Metropolis (SA), ewolucyjny, genetyczny (GA), przeszukiwania lokalnego Solis & Wets (LS), hybrydowy algorytm Lamarckiana (LGA).
Funkcja oceniająca konfiguracje układu ligand-receptor powinna możliwie dokładnie estymować energię swobodną tworzenia kompleksu ligand-receptor. Zależność energii swobodnej Gibbsa od stałej równowagi dwóch stanów termodynamicznych pozwala na orientacyjne wyznaczenie stałych inhibicji dokowanych ligandów:
Funkcja energii swobodnej powinna zatem uwzględniać następujące stany układu termodynamicznego (L-ligand, P-białko, V - energia potencjalna, ΔS - zmiana entropii):
Bardzo często estymatory energii swobodnej łączą elementy mechaniki molekuarnej z parametrami empirycznymi. Przykładem może być semiempiryczna energia swobodna wiązania pola siłowego AutoDock: