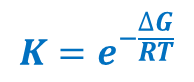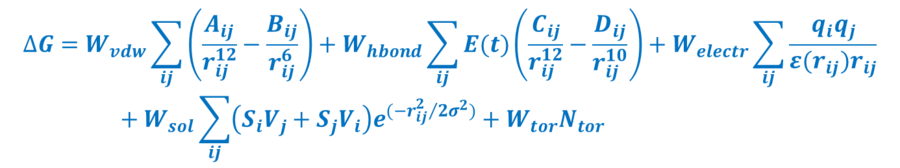m (→Dokowanie molekularne) |
m (→Dokowanie molekularne) |
||
| Linia 1: | Linia 1: | ||
==Dokowanie molekularne== | ==Dokowanie molekularne== | ||
| − | '''Dokowanie molekularne''' jest dziedziną modelowania molekularnego, która polega na poszukiwaniu optymalnego (o najniższej energii) modu wiązania liganda w strukturze receptora (oddziaływania liganda z receptorem). Zbiór wszystkich konfiguracji liganda i receptora tworzy przestrzeń konformacyjną z charakterystycznym dla siebie układem powierzchni energii potencjalnej (w przyjętym polu siłowym). [[Image:Dokowanie-Rysunek1.png|thumb|right|450px|Rysunek 1: Mody wiązania ligandów w kieszeni ATP kinazy Rio1 zidentyfikowane w oparciu o dokowanie molekularne.{{r|Mielecki et al. 2013}}]] Dokowanie molekularne poszukuje '''minimum globalnego''' pola siłowego wyznaczonego dla kompleksu ligand-receptor. Realizacja tego celu, a więc obliczeniowe rozwiązanie problemu dokowania, wymaga opracowania dwóch głównych metod: '''algorytmu przeszukiwania''' wielowymiarowej przestrzeni konfiguracyjnej, a także '''funkcji oceniającej''' (scoring function) zaproponowane konfiguracje układu ligand-receptor. Ligand odnosi się najczęściej do związku niskocząsteczkowego, natomiast receptor - do białka. Dokowanie może jednak dotyczyć różnych par makromolekuł, np. tworzenia kompleksów białko-białko. | + | '''Dokowanie molekularne''' jest dziedziną modelowania molekularnego, która polega na poszukiwaniu optymalnego (o najniższej energii) modu wiązania liganda w strukturze receptora (oddziaływania liganda z receptorem). Zbiór wszystkich konfiguracji liganda i receptora tworzy przestrzeń konformacyjną z charakterystycznym dla siebie układem powierzchni energii potencjalnej (w przyjętym polu siłowym). [[Image:Dokowanie-Rysunek1.png|thumb|right|450px|Rysunek 1: Mody wiązania ligandów w kieszeni ATP kinazy Rio1 zidentyfikowane w oparciu o dokowanie molekularne.{{r|Mielecki et al. 2013}}]] Dokowanie molekularne poszukuje '''minimum globalnego''' pola siłowego wyznaczonego dla kompleksu ligand-receptor. Realizacja tego celu, a więc obliczeniowe rozwiązanie problemu dokowania, wymaga opracowania dwóch głównych metod: '''algorytmu przeszukiwania''' wielowymiarowej przestrzeni konfiguracyjnej, a także '''funkcji oceniającej''' (scoring function) zaproponowane konfiguracje układu ligand-receptor. ''Ligand'' odnosi się najczęściej do związku niskocząsteczkowego, natomiast ''receptor'' - do białka. Dokowanie może jednak dotyczyć różnych par makromolekuł, np. tworzenia kompleksów białko-białko. |
Dokowanie molekularne może być pomocne w: | Dokowanie molekularne może być pomocne w: | ||
Wersja z 20:18, 22 lis 2014
Dokowanie molekularne
Dokowanie molekularne jest dziedziną modelowania molekularnego, która polega na poszukiwaniu optymalnego (o najniższej energii) modu wiązania liganda w strukturze receptora (oddziaływania liganda z receptorem). Zbiór wszystkich konfiguracji liganda i receptora tworzy przestrzeń konformacyjną z charakterystycznym dla siebie układem powierzchni energii potencjalnej (w przyjętym polu siłowym).
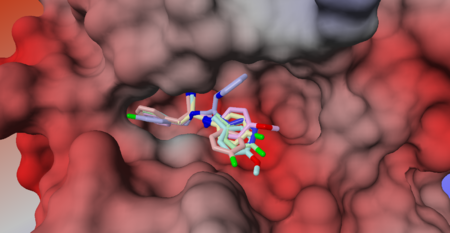
Dokowanie molekularne poszukuje minimum globalnego pola siłowego wyznaczonego dla kompleksu ligand-receptor. Realizacja tego celu, a więc obliczeniowe rozwiązanie problemu dokowania, wymaga opracowania dwóch głównych metod: algorytmu przeszukiwania wielowymiarowej przestrzeni konfiguracyjnej, a także funkcji oceniającej (scoring function) zaproponowane konfiguracje układu ligand-receptor. Ligand odnosi się najczęściej do związku niskocząsteczkowego, natomiast receptor - do białka. Dokowanie może jednak dotyczyć różnych par makromolekuł, np. tworzenia kompleksów białko-białko.
Dokowanie molekularne może być pomocne w:
- projektowaniu nowych ligandów działających jak inhibitory (potencjalne leki),
- przeszukiwaniu baz danych związków niskocząsteczkowych, które oddziałują z danym receptorem (białkiem lub kwasem nukleinowym),
- przewidywaniu modu wiązania związków niskocząsteczkowych w strukturze receptora (białko, kwas nukleinowy), badaniu mechanizmu działania znanych inhibitorów.
Wielkość przestrzeni konfiguracyjnej, a więc możliwych stanów, jakie może przyjąć układ liganda oddziałującego z receptorem (np. białko, kwas nukleinowy) rośnie wykładniczo wraz z liczbą wymiarów D (liczbą stopni swobody układu: liganda i receptora). Jeśli przyjmiemy taki sam stopień skomplikowania - możliwych wartości N dla każdego z wymiarów, to wielkość przestrzeni konfiguracyjnej (konformacyjnej) będzie równa:
Wymiarami mogą być np.: położenie liganda, orientacja cząsteczki liganda czy swobodne wiązanie w cząsteczce liganda (z możliwością rotacji). Pełna swoboda konformacyjna receptora powoduje znaczący wzrost wielkości przestrzeni konformacyjnej ze względu na wielkość jego cząsteczki. Często zatem ogranicza się pełną swobodę konformacyjną do cząsteczki liganda w dokowaniu tzw. rigid-flexible lub też do liganda i wybranych reszt aminokwasowych w kieszeni wiążącej białka.
Ze względu na znaczny koszt obliczeniowy systematycznego i wyczerpującego przeszukania przestrzeni konfiguracyjnej, algorytmy przeszukania opracowane dla potrzeb dokowania molekularnego to najczęściej algorytmy heurystyczne i stochastyczne. Algorytmy te próbkują przestrzeń konfiguracyjną w sposób losowy (w przeciwieństwie do algorytmów deterministycznych i systematycznych). W związku z tym proponują one przybliżone rozwiązanie problemu, które nie musi być rozwiązaniem globalnym, optymalnym. Wynik dokowania nie musi być optymalny. Przykładowe algorytmy przeszukiwania to: symulowane wyżarzanie typu Metropolis (SA), ewolucyjny, genetyczny (GA), przeszukiwania lokalnego Solis & Wets (LS), hybrydowy algorytm Lamarckiana (LGA).
Funkcje oceniające mogą być oparte na wyrażeniach fizycznych mechaniki molekularnej lub wyrażeniach empirycznych. Funkcje empiryczne mogą np. wykorzystywać algorytmy geometrycznego dopasowania liganda do receptora (np. pierwsze algorytmy programu DOCK). Atomistyczna funkcja oceniająca konfiguracje układu ligand-receptor powinna jednakże możliwie dokładnie estymować energię swobodną tworzenia kompleksu ligand-receptor. Energia swobodna Gibbsa zależy od stałej równowagi dwóch stanów termodynamicznych w następujący sposób:
Funkcja energii swobodnej powinna uwzględniać następujące stany układu termodynamicznego (L-ligand, P-białko, V - energia potencjalna, ΔS - zmiana entropii):
W przypadku dokowania flexible-rigid, człony powyższego równania związane z energią potencjalną receptora (białka) w konfiguracji niezwiązanej i związanej z ligandem znoszą się wzajemnie. Bardzo często estymatory energii swobodnej łączą elementy mechaniki molekuarnej z parametrami empirycznymi. Przykładem może być semiempiryczna energia swobodna wiązania pola siłowego AutoDock:
gdzie:
- ΔGVDW - fizyczne wyrażenie na potencjał Lenarda-Jonesa;
- ΔGhbond - fizyczne wyrażenie na potencjał tworzenia ukierunkowanych wiązań wodoworwych;
- ΔGelectr - fizyczne wyrażenie na potencjał Coulomba;
- ΔGdesolv - empiryczne wyrażenie na potencjał desolwatacji;
- ΔGtor - empiryczne wyrażenie na zminę entropii tworzenia kompleksu ligand-receptor (Ntor to liczba stopni swobody liganda);
- współczynniki W to empiryczne parametry skalujące.
Metodologia dokowania molekularnego to kompromis między dokładnością a kosztem obliczeniowym związanym z wielkością przestrzeni konfiguracyjnej. Pozwala ono w stosunkowo krótkim czasie wyznaczyć prawdopodobne mody oddziaływania liganda z receptorem, które w wielu przypadkach nie odbiegają znacznie od struktur krystalograficznych. Manipulowanie dokładnością przeszukiwania przestrzeni konfiguracyjnej, a więc kosztem obliczeniowym, sprzyja zastosowaniu dokowania molekularnego do wirtualnego screeningu dużych bibliotek związków niskocząsteczkowych. Wyniki dokowania mogą być weryfikowane z zastosowaniem metod eksperymentalnych (np. inhibicja aktywności enzymatycznej, krystalografia, wygaszanie fluorescencyjne) lub też obliczeniowych (np. dynamika molekularna).
Nie zawsze jednak możliwe jest przewidywanie optymalnego modu oddziaływania liganda z receptorem, a wynik dokowania obarczony jest znacznym błędem. Wiąże się to głównie z niewystrczającym stopniem dokładności (szczegółowości) funkcji oceniających i estymacją energii swobodnej. Inne ograniczenia dokowania molekularnego to:
- wymaga struktury eksperymentalnej receptora (białka)
- zbyt duże zmiany konformacyjne receptora (białka) indukowane wiązaniem liganda
- zbyt wysoka liczba stopni swobody liganda
- nie uwzględnia oddziaływań, w których mogą pośredniczyć cząsteczki rozpuszczalnika.
Szereg strategii pozwala poprawić dokładność dokowania molekularnego. Przede wszystkim rosnąca liczba eksperymentalych struktur kompleksów, dla których znane są stałe oddziaływania (inhibicji), pozwala na optymalizację parametryzacji funkcji oceniających. Możliwe jest również dodawanie cząsteczek wody w przypadku, gdy wyniki innych doświadczeń wskazują na to, iż cząsteczki te mogą pośredniczyć w oddziaływaniu liganda z receptorem. Rozwijane są ponadto strategie oparte na dokowaniu do zespołu konformacyjnego receptora wygenerowane z zastosowaniem metod mechaniki czy dynamiki molekularnej, np. w oparciu o biblioteki rotamerów reszt aminokwasowych w białku.
Literatura
- ↑ Mielecki M., Krawiec K., Kiburu I., Grzelak K., Zagórski W., Kierdaszuk B., Kowa K., Fokt I., Szymanski S., Świerk P., Szeja W., Priebe W., Lesyng B., LaRonde-LeBlanc N.. Development of novel molecular probes of the Rio1 atypical protein kinase. Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 2013;1834(7):1292-1301. doi:10.1016/j.bbapap.2013.03.012. Błąd rozszerzenia cite: Nieprawidłowy znacznik
<ref>; nazwę „Mielecki et al. 2013” zdefiniowano więcej niż raz z różną zawartością Błąd rozszerzenia cite: Nieprawidłowy znacznik<ref>; nazwę „Mielecki et al. 2013” zdefiniowano więcej niż raz z różną zawartością
Błąd rozszerzenia cite: Znacznik <ref> o nazwie „Huey et al. 2007”, zdefiniowany w <references>, nie był użyty wcześniej w treści.
Błąd rozszerzenia cite: Znacznik <ref> o nazwie „Morris et al. 2013”, zdefiniowany w <references>, nie był użyty wcześniej w treści.
Błąd rozszerzenia cite: Znacznik <ref> o nazwie „Morris et al. 2009”, zdefiniowany w <references>, nie był użyty wcześniej w treści.
Błąd rozszerzenia cite: Znacznik <ref> o nazwie „Goodsell et al. 1996”, zdefiniowany w <references>, nie był użyty wcześniej w treści.
`